David Baker是美国华盛顿大学的教授,结构生物学家,2024年诺贝尔化学奖的获奖者之一,主攻蛋白质折叠、蛋白质设计以及生物大分子结构解析等领域。其实验室团队仅2024年在全新蛋白设计领域发表了近19篇研究成果,通过人工智能和计算建模的结合,推动了从头设计蛋白的技术革命。他们展示了如何通过深度学习算法、分子动力学模拟和实验验证,快速、高效地构建功能多样的蛋白,解决了许多传统方法无法攻克的科学难题。
一、研究亮点
此次在bioRxiv上发表的《De novo design of miniprotein agonists and antagonists targeting G protein-coupled receptors》研究针对的是药物发现与开发的核心靶点G蛋白偶联受体(GPCRs)。GPCR靶点因其多穿膜蛋白且具有构象动态性,设计其蛋白激动剂和拮抗剂一直存在挑战。这里整体概括了该项研究的设计思路及筛选方法。
- 该研究结合计算机与实验的设计方法,用于开发具有高亲和力、高效率、高选择性的GPCR靶向性微蛋白。该方法结合了RFdiffusion算法、MetaGen框架及基于受体转向 (RD)显微镜分析的高通量筛选技术。
- 该研究采用两种设计策略
基序导向的RF diffusion:靶向受体特定热点区域,实现深位点作用。
MetaGen:利用从Alpha Fold衍生的宏蛋白质组结构支架生成多样化设计。
- 受体转向显微镜分析(RD)筛选平台可直接在人体细胞内运行,无需受体纯化步骤,确保与复杂的细胞环境兼容。该技术可高效筛选多达100,000种设计方案。
- 设计的微蛋白激动剂成功激活了与瘙痒和疼痛相关的GPCR-MRGPRX1,其中筛选出来的两个结合蛋白表现出完全激动和部分激动活性,冷冻电镜结构数据进一步验证了其结合精准性和作用机理。
- 该研究还开发了靶向CXCR4、GLP1R、GIPR、GCGR和CGRPR的拮抗剂,其亲和力在纳摩尔至皮摩尔水平。其中CGRPR拮抗剂dC2_049表现出卓越的选择性和效力。
- 冷冻电镜结构分析证实,设计的拮抗剂可通过空间位阻封闭结合位点并阻断激活,为GPCR靶向治疗提供了新的机遇。
- 计算机设计与高通量筛选的成功结合,为开发靶向GPCR的新型蛋白质疗法提供了一条充满希望的途径。
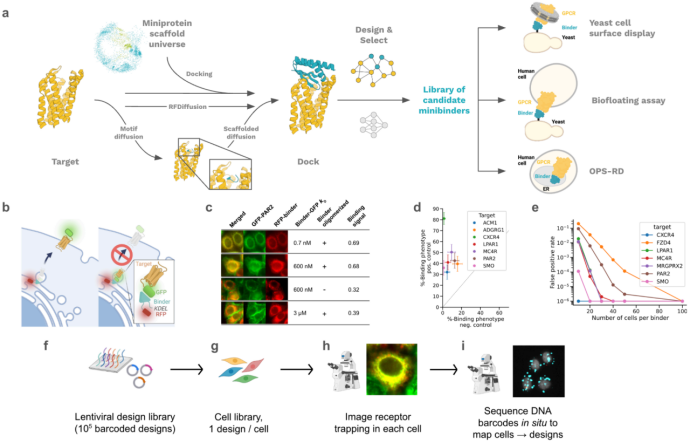
Fig. 1. GPCR binder computational design and screening methods [1]
二、缔码生物CXCR4-nanodisc助力
缔码生物CXCR4-nanodisc (FLP100074)参与了这项研究的实验部分,用于酵母展示及筛选。

缔码生物专为全长膜蛋白打造的Nanodisc解决方案,解决了多穿膜蛋白难表达、难定量、难溶解、难纯化等问题,可使跨膜蛋白处于天然膜环境中,保持生物活性。而且后续实验中不需要添加洗涤剂,可以直接应用于细胞学分析。另外,基于哺乳动物细胞表达系统的翻译后修饰保证,这也为跨膜蛋白的功能提供了进一步保障。目前,缔码生物已利用Nanodisc开发出了500+种跨膜蛋白现货产品,靶点涵盖GPCR家族和离子通道家族,最高的跨膜次数可达24次。
参考文献:
[1] https://doi.org/10.1101/2025.03.23.644666


