在蛋白标记、抗体偶联及多功能融合蛋白构建等研究与药物开发场景中,位点特异性蛋白连接技术正变得越来越重要[1]。其中,来源于革兰氏阳性菌的转肽酶Sortase A(SrtA)因能够特异识别LPXTG基序并催化与多聚甘氨酸底物的定点连接,已成为应用最广泛的生物偶联工具之一。随着应用复杂度不断提升,研究者也开始通过工程化改造与定向进化来提升Sortase A的催化效率和反应性能,从而满足更高效、更可控的蛋白偶联需求。
一、什么是Sortase A蛋白酶?
Sortase A蛋白酶是一类天然存在的转肽酶,能够识别并作用于特定的短肽序列,最典型的是LPXTG识别序列[2,3]。在温和的反应条件下,Sortase A可识别蛋白中的特定短肽序列LPXTG,并在Thr(T)与Gly(G)之间进行精确切割。其中X表示任意氨基酸,该位点对整体识别影响较小。切割后形成的酶-底物中间体可进一步与N端带有寡聚Gly(通常为 GGG)的分子发生转肽反应,从而实现新肽段或功能模块在该位点的定点连接。与依赖化学活化的随机修饰方法不同,Sortase A的反应具有高度序列特异性,产物结构清晰、可预测,尤其适用于对构象和功能高度敏感的蛋白体系。
因此,Sortase A是一种能够在预定义位点实现蛋白精准处理的转肽酶工具。
二、应用一:Sortase A在标签去除中的应用
2.1 为什么需要酶法标签去除?
在重组蛋白的表达与纯化过程中,各类亲和标签(如His-tag、Flag-tag等)是不可或缺的工具。然而,这些标签的存在本身并非最终目的。在获得高纯度目标蛋白后,标签残留可能对后续研究造成干扰,例如:
- 改变蛋白的天然构象
- 影响生物活性或结合能力
- 干扰后续的定点标记、偶联或功能评价实验
因此,在功能蛋白制备阶段,安全、可控地去除融合标签,是保证实验质量的重要环节。
2.2 Sortase A作为标签去除工具的优势
利用Sortase A进行位点特异性切割,可在预先设计的识别序列处精准去除标签,具有以下优势[4]:
- 切割位点清晰,不引入多余残基
- 反应条件温和,适合结构和功能敏感蛋白
- 产物高度可预测,有利于下游质量控制
值得注意的是,Sortase A在标签去除中所体现的“位点可控”特性,同样为其在后续蛋白定点修饰中的应用奠定了基础。
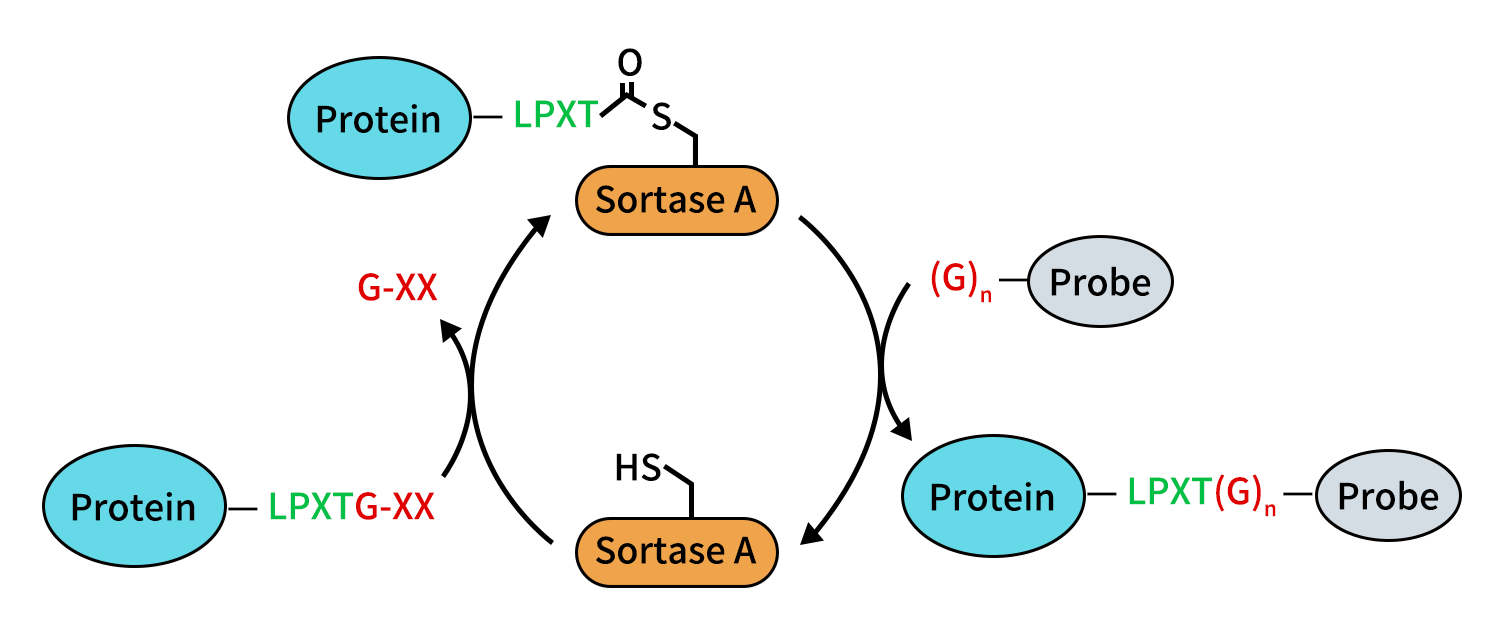
Figure 1. N-terminal labeling of proteins.
三、应用二:Sortase A介导的位点特异性蛋白标记与偶联
3.1 为什么定点标记正在取代随机标记?
在蛋白功能研究和检测体系中,标记分子的引入往往不可避免。与传统随机标记相比,定点标记在以下方面表现出显著优势[5,6]:
- 避免关键功能区域被过度修饰
- 显著提高实验重复性和数据一致性
- 更适合对构象和活性高度敏感的蛋白
随着研究精度要求的不断提高,定点标记已成为高质量蛋白工程项目的首选策略。
3.2 Sortase A在定点标记中的核心优势
基于Sortase A的定点偶联体系,研究者可以在预设位置、以统一方向引入标记分子,其核心优势包括[5-7]:
- 标记位点与连接方向可控
- 连接方式高度一致,降低样品异质性
- 兼容体积较大或结构复杂的标记物
3.3 Sortase A在抗体定点偶联与ADC构建中的应用
在抗体相关的偶联应用中,尤其是抗体-药物偶联物(Antibody-Drug Conjugates, ADCs)领域,偶联位点的可控性直接决定了产品的均一性、稳定性和体内行为。传统基于赖氨酸或半胱氨酸的随机偶联策略,往往会产生药物负载数(DAR)分布宽、构象异质性高的问题,从而影响疗效与安全性。
通过在抗体重链或轻链末端引入LPXTG识别序列,Sortase A可在不干扰抗原结合位点的前提下,实现抗体的位点特异性偶联,用于连接小分子细胞毒药物,药物连接子(linker)和荧光探针或示踪分子。该策略能够获得定义明确的偶联位点与可控的DAR值,显著提升ADC分子的批次一致性和可预测性。目前,Sortase A介导的抗体定点偶联已被广泛用于ADC构建、机制研究以及候选分子筛选阶段,为下一代高均一性ADC的开发提供了重要工具基础。
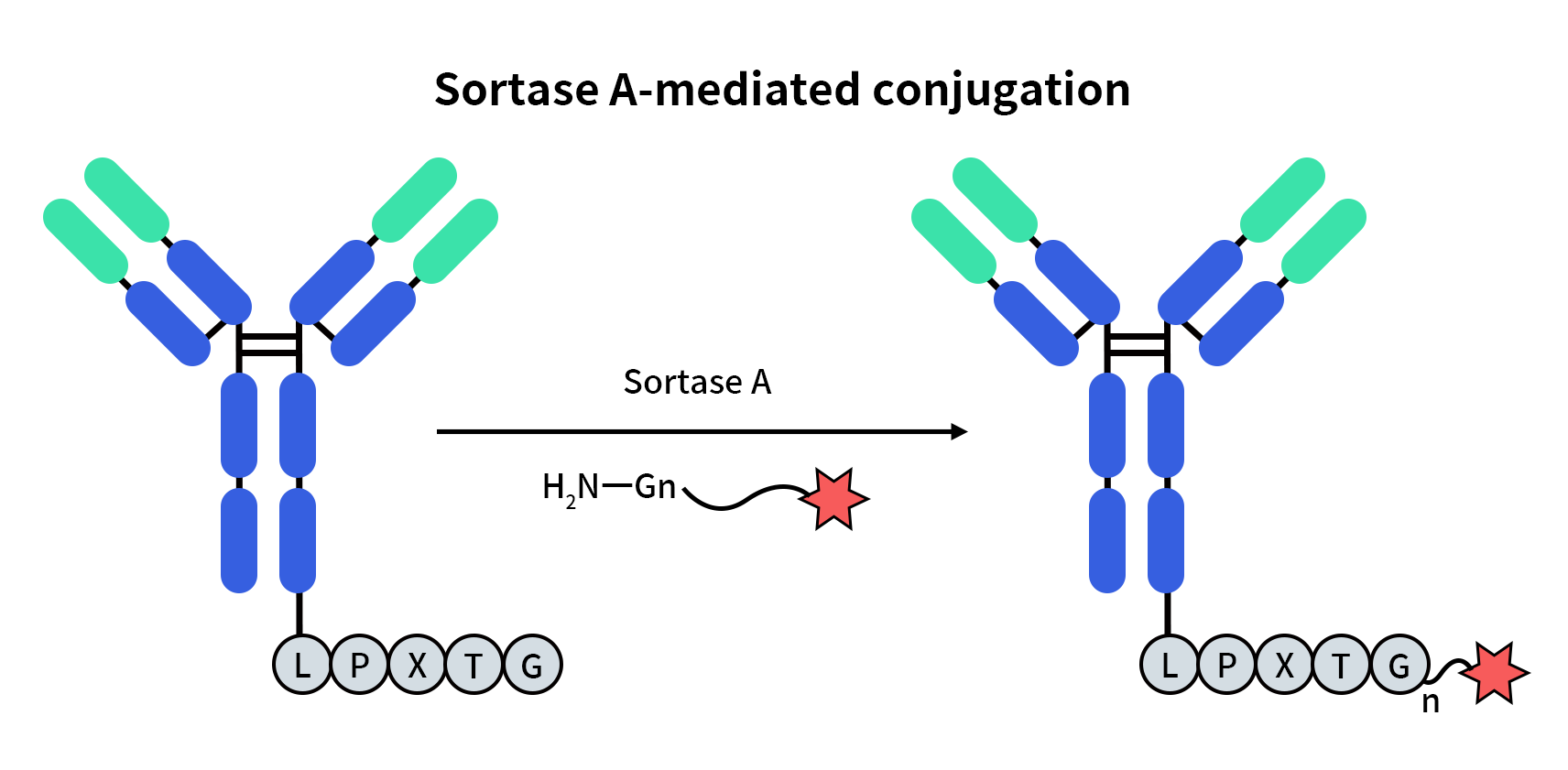
Figure 2. Sortase A-mediated conjugation.
四、Sortase A 5M:为位点特异性蛋白工程优化的改造酶
随着位点特异性蛋白工程对效率和体系兼容性的要求不断提高,未经工程化改造的野生型Sortase A在复杂蛋白体系中逐渐暴露出反应速率受限、对大分子底物适应性不足等问题。尤其是在抗体、Fc融合蛋白以及大体积标记物参与的应用场景中,更高活性、更稳定的工程化酶变体成为提升实验可控性和成功率的关键。
Sortase A 5M是由David R.Liu教授团队通过定向进化策略获得的高活性工程化变体,包含五个关键突变(P94R、D160N、D165A、K190E、K196T),在保持LPXTG序列识别特异性的同时,显著提升了转肽反应效率。与野生型酶相比,该变体在较温和条件下即可实现更高效的连接反应,并对Fc融合蛋白、抗体及大分子功能标签表现出更好的底物兼容性[8]。在实际应用中,其优势主要体现在以下几个方面:
l首先,转肽效率更高,在较低酶用量和较短反应时间下即可获得稳定、可重复的连接产物;
l其次,反应稳定性更好,对缓冲体系、底物浓度变化具有更高耐受性,更适合标准化和规模化操作;
第三,对复杂或体积较大的底物具有更好的兼容性,包括Fc融合蛋白、抗体分子以及大分子荧光或功能标签。
缔码提供的Sortase A 5M产品基于这一经过充分验证的工程化酶体系,可稳定支持标签去除、定点生物素化、荧光蛋白偶联及抗体相关定点修饰等多类应用。下文所展示的Biotin与PE定点标记案例,均建立在Sortase A 5M体系之上,并已完成内部功能验证。
五、缔码基于Sortase A 5M的验证应用:Biotin与PE标记
5.1 Sortase A 5M介导的Biotin定点标记
在Biotin标记体系中,随机生物素化往往会导致结合位点受损或信号不稳定。通过Sortase A 5M实现位点特异性Biotinylation,可有效避免上述问题。该策略已在内部应用中得到验证,适用于多种下游检测平台,包括:ELISA,BLI/SPR动力学分析和Pull-down等相互作用研究体系。
案例1:我们选取BCMA-hFc融合蛋白作为模型分子,利用Sortase A 5M介导的C端定点偶联体系,在其C端引入生物素标签,并通过ELISA方法系统评估偶联前后蛋白的抗原结合活性保留情况,以验证该策略在抗体及免疫相关蛋白功能化应用中的适用性与可靠性。
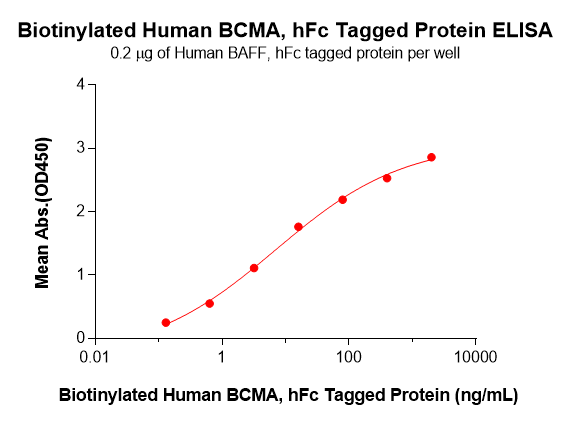
Figure 3. Biotinylated Human BCMA-hFc shows dose-dependent binding to immobilized Human BAFF-hFc, as detected by HRP–streptavidin, with an EC50 of 7.03 ng/mL.
案例2:我们以B7-H3的单克隆抗体作为模型,通过 Sortase A 5M介导的C端定点生物素化策略,对抗体进行精准功能化修饰,并进一步将其应用于Sandwich ELISA体系中,以评估该定点标记方式对抗体检测性能与功能保留的影响。
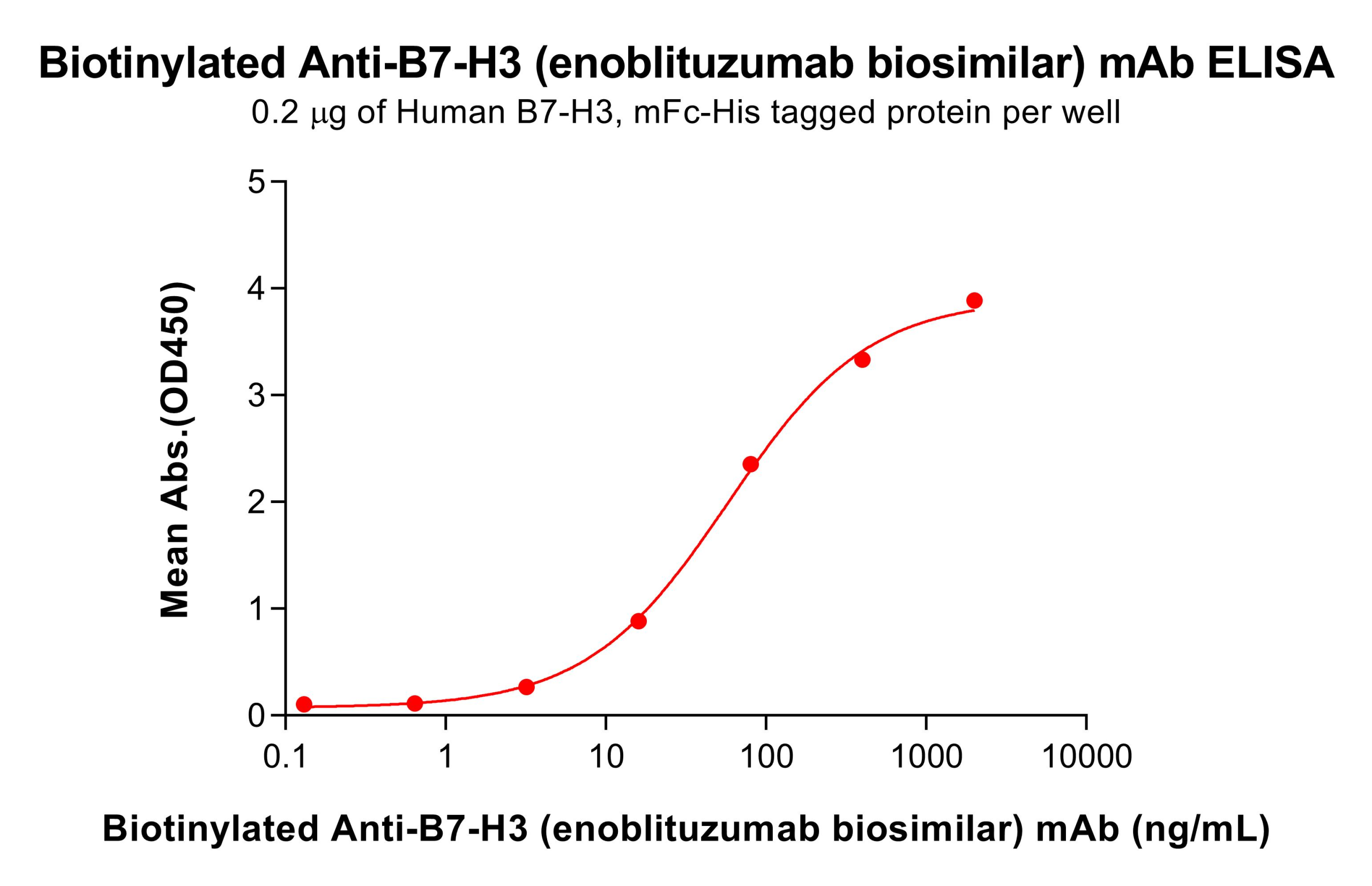
Figure 4. Biotinylated anti-B7-H3 mAb (enoblituzumab biosimilar) exhibits linear binding to ELISA-immobilized Human B7-H3 mFc-His within 16-80 ng/mL.
5.2 Sortase A 5M介导的PE定点标记
PE(Phycoerythrin)属于体积较大的荧光标记物,对偶联位点和空间构象极为敏感。随机偶联不仅可能削弱荧光性能,还可能影响蛋白的天然结构。通过Sortase A 5M实现PE的定点偶联,可在明确位点、统一方向下完成连接,兼顾荧光性能与蛋白功能完整性。该方案同样已完成内部应用验证,特别适用于流式细胞术等高灵敏检测场景。
案例1:为评估Sortase A 5M介导的PE定点标记是否适用于流式细胞术等对构象和位阻高度敏感的应用场景,我们进一步在细胞水平对标记后的抗B7-H3抗体进行功能验证。
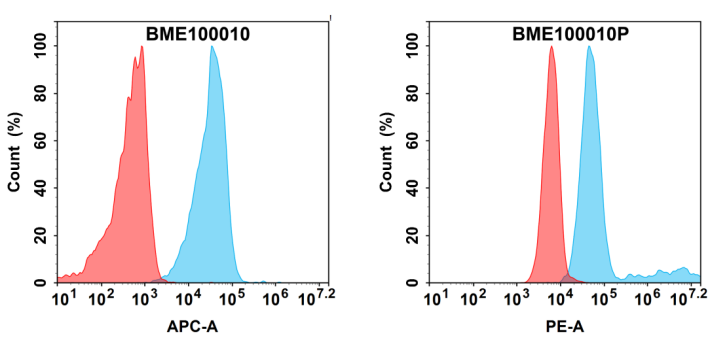
Figure 5. Flow cytometry comparison of Anti-B7-H3 (enoblituzumab biosimilar) mAb before and after PE conjugation on HEK293 cells. Blue: Anti-B7-H3; Red: isotype control.
六、Sortase A不只是“定点偶联酶”
从技术本质来看,Sortase A并非单一用途的定点偶联工具,而是一个位点特异性蛋白工程平台。既可以用于去除不需要的标签,也可以在指定位置引入功能模块或标记分子,其核心价值在于对蛋白结构与功能的精准编辑能力,为复杂蛋白体系的设计与优化提供了高度可控的技术路径。
综上所述,Sortase A本质上是一种用于位点特异性蛋白工程的通用酶工具,而不仅仅是定点偶联的辅助手段。为进一步支持蛋白工程研究与功能化应用,我们提供高质量Sortase A蛋白,可广泛应用于标签去除、位点特异性标记以及抗体或Fc融合蛋白的定点偶联。结合我们丰富的应用验证案例(如BCMA-hFc、BM35抗体、B7-H3抗体等),使用该Sortase A蛋白可显著提升实验可控性、重复性和数据可靠性。
如需定制或获取更多技术支持,欢迎致电咨询。(400-006-0995/18062749453)
参考文献
- Popp, M. W., & Ploegh, H. L. (2011). Making and breaking peptide bonds: protein engineering using sortase. Angewandte Chemie International Edition, 50(22), 5024–5032.
- Mazmanian, S. K., Liu, G., Ton-That, H., & Schneewind, O. (1999). Staphylococcus aureus sortase, an enzyme that anchors surface proteins to the cell wall. Science, 285(5428), 760–763.
- Ton-That, H., Mazmanian, S. K., Alksne, L., & Schneewind, O. (2002). Anchoring of surface proteins to the cell wall of Staphylococcus aureus. Journal of Biological Chemistry, 277(9), 7447–7452.
- Witte, M. D., Cragnolini, J. J., Dougan, S. K., Yoder, N. C., Popp, M. W., & Ploegh, H. L. (2012). Preparation of unnatural N-to-N and C-to-C protein fusions. Proceedings of the National Academy of Sciences, 109(30), 11993–11998.
- Antos, J. M., Chew, G.-L., Guimaraes, C. P., Yoder, N. C., Grotenbreg, G. M., Popp, M. W., & Ploegh, H. L. (2009). Site-specific protein labeling via sortase-mediated transpeptidation. Current Protocols in Protein Science, Chapter 15, Unit 15.3.
- Theile, C. S., et al. (2013). Site-specific N-terminal labeling of proteins using sortase-mediated reactions. Nature Protocols, 8(9), 1800–1807.
- Guimaraes, C. P., Carette, J. E., Varadarajan, M., Antos, J., Popp, M. W., Spooner, E., Brummelkamp, T. R., & Ploegh, H. L. (2011). Identification of host cell factors required for intoxication through use of modified cholera toxin. The Journal of cell biology, 195(5), 751–764.
- Chen, B. M. Dorr & D. R. Liu, A general strategy for the evolution of bond-forming enzymes using yeast display, Proc. Natl. Acad. Sci. U.S.A., 108(28):11399–11404 (2011).


